Inserm Bio-Santé Toulouse
IBST
Inserm Bio-Santé Toulouse (IBST) est composé de deux plateformes Inserm situées dans deux Instituts de Recherche de Toulouse (I2MC et CRCT) pour un total de 4 ingénieurs. Depuis 2017, ces plateformes accompagnent les utilisateurs depuis la conception des expériences single-cell jusqu’à l’analyse des données, en passant par la génération des librairies et le séquençage. La plateforme propose toutes les solutions et protocoles commercialisés par 10x Genomics, avec une forte expertise en expression génique, CITE-seq, profilage immunitaire (TCR, BCR) et transcriptomique spatiale.
ÉQUIPE
Expertises
ÉQUIPEMENTS
CONTACT
L’Équipe

Emeline LHUILLIER

Carine VALLE

Emeline SAROT
EXPERTISES
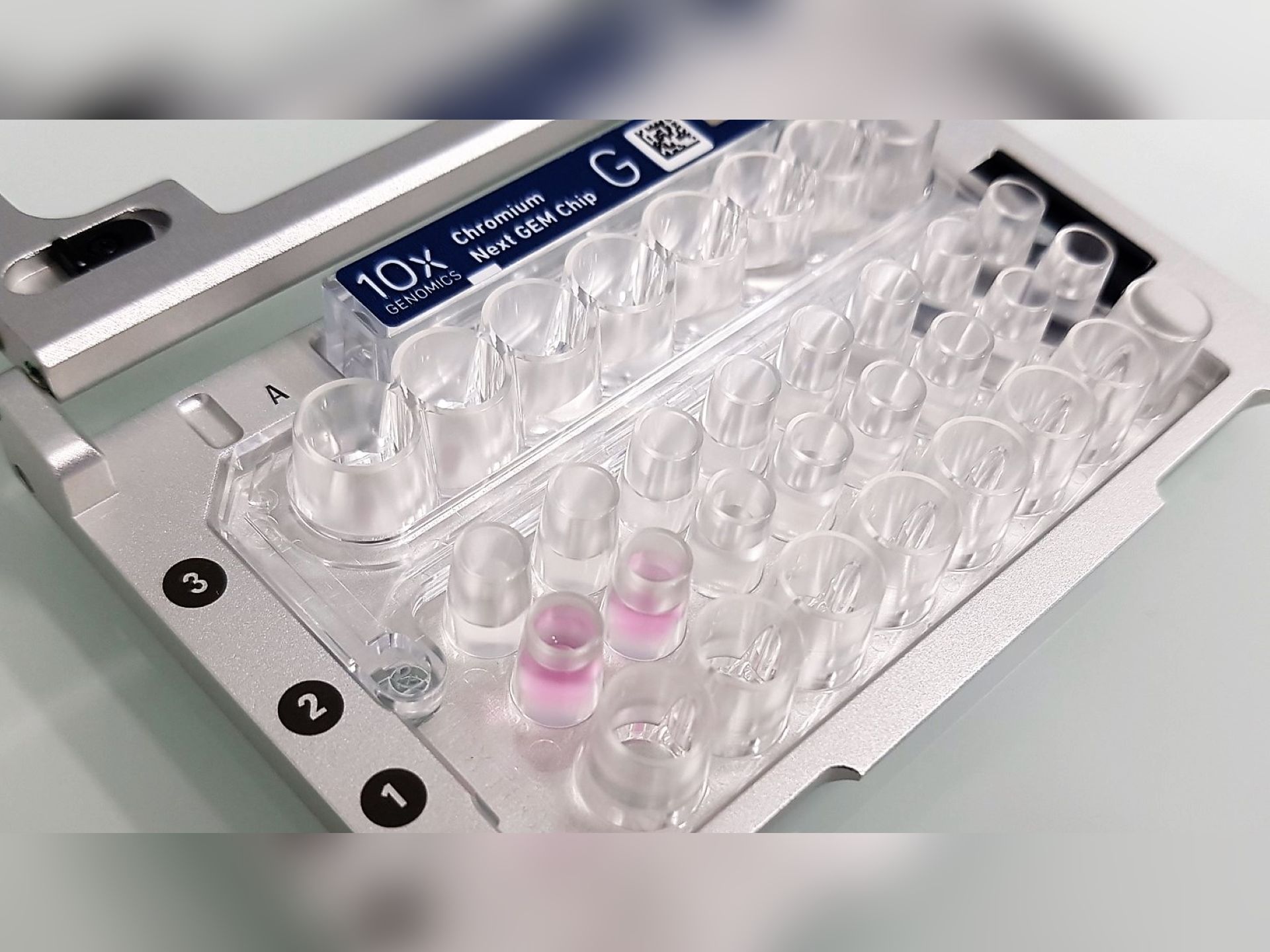
SINGLE CELL
« Chromium Single Cell Gene Expression » fournit l’expression des gènes en 3′ sur cellule unique, ainsi que des capacités multiomiques pour profiler des dizaines de milliers de cellules. Cette technologie permet d’explorer l’hétérogénéité et la complexité de la diversité cellulaire, les nouvelles cibles et les biomarqueurs avec l’expression combinée des gènes et des protéines de surface dans chaque cellule.
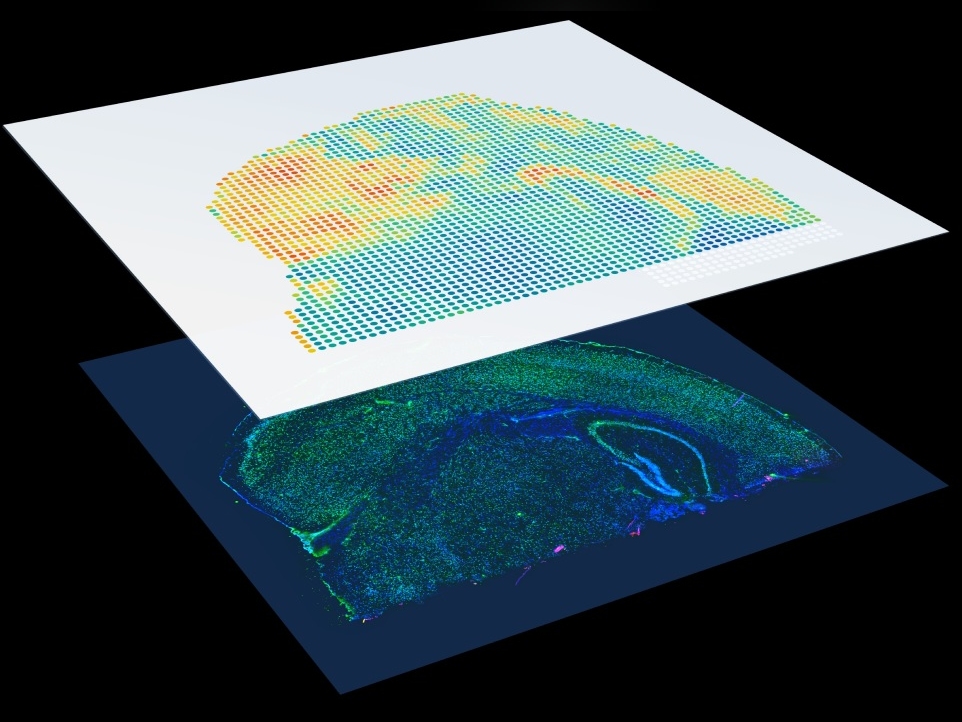
SPATIAL OMIC
« Visium Spatial Gene Expression » est une solution de profilage moléculaire de nouvelle génération pour la classification des tissus basée sur l’ARNm total. Cette technologie permet de cartographier l’ensemble du transcriptome avec le contexte morphologique dans des tissus FFPE ou fraîchement congelés pour découvrir de nouvelles perspectives sur le développement normal, pathologique, ou en recherche clinique translationnelle.

Séquençage NGS
RNAseq, ChIPseq, WGS… : accompagnement dans le design des projets, construction des librairies, coordination avec les plateformes de séquençage et d’analyses bioinfo.
ÉQUIPEMENTS

2 Chromium Single Cell Controllers
Le Chromium Controller utilise une microfluidique avancée pour effectuer le partitionnement et le codage à barres de cellules uniques en quelques minutes. Alimenté par la technologie Next GEM, le Chromium Controller permet l’analyse intégrée de cellules uniques à grande échelle.

NextSeq 550
La principale application proposée est l’analyse du transcriptome (RNA-Seq) codant après sélection polyA ou total par déplétion ribosomique ou en cellule unique. L’appareil a également les capacités pour gérer des projets de séquençage des petits ARNs (miRNAs), d’amplicon ou de panel, d’exome et de ChIP (immunoprécipitation de la chromatine). La mise en place de protocole pour ces applications se fait au fur et à mesure des demandes et des spécifications de chaque projet.

NovaSeq6000
Le NovaSeq 6000 ouvre une nouvelle ère dans le séquençage avec des innovations révolutionnaires, offrant aux utilisateurs le débit, la vitesse et la flexibilité nécessaires pour réaliser des projets plus rapidement et plus économiquement que jamais. Tirant parti de la technologie éprouvée de séquençage de nouvelle génération (NGS) d’Illumina, de plusieurs types de Flow Cell, de deux modes de chargement de librairies et de diverses combinaisons de longueurs de lecture, le système NovaSeq 6000 permet un dimensionnement optimal du débit pour répondre à pratiquement tous les besoins d’étude.
CONTACT
N’hésitez pas à nous contacter pour vos projets.
liste.ibst@inserm.fr


Inserm/UPS UMR 1297 - I2MC Institut des Maladies Métaboliques et Cardiovasculaires
1 avenue Jean Poulhès - BP 84225 - 31432 Toulouse Cedex 4
Tél. : 05 61 32 56 00
Horaires
Du lundi au vendredi
8h30 - 12h30 / 13h45 -16h45



